Dank eines Stipendiums im Rahmen der Auricher Wissenschaftstage war es mir möglich, ein zweiwöchiges Praktikum (11.10-23.10.2020) am Institut für Strukturbiologie in Bonn zu absolvieren.
Mein Praktikum startete am Montagmorgen um 09 Uhr mit dem sogenannten „Labmeeting“. Dort wurden Pläne und Ziele für die kommende Woche vorgestellt und besprochen. Meist ging es dabei um organisatorische Fragen, wie meine Vorstellung in der Arbeitsgruppe. Aufgrund der akuten Pandemie-Situation mussten auch Regeln bzgl. des Infektionsschutzes besprochen werden. Nachdem alle Punkte abgearbeitet waren, teilte sich das Team in mehrere Arbeitsgruppen auf und ich konnte einem sehr spannenden Vortrag von Dr. Jonas Möcking beiwohnen, der über Proteininteraktion referierte.
Anschließend begann für mich die Arbeit im Labor. In meiner ersten Woche habe ich überwiegend mit Anja Kopp und Martin Peters gearbeitet. Zwischen meinen Praxisphasen durfte ich bei allen Mitarbeitern über die Schulter schauen und Fragen stellen, die mir immer ausführlich beantwortet wurden.
Die Strukturbiologie beschäftigt sich mit der Struktur von Proteinen und damit, wie molekulare Veränderungen sich auf die Arbeit bzw. Interaktion dieser auswirken. Grundsätzlich lässt sich die Herstellung von Proteinen in drei Schritte einteilen. Als erstes erfolgt die Klonierung, dann die Expression und anschließend die Aufreinigung. Wenn alle Schritte erfolgreich absolviert worden sind, kann mit den entstandenen Proteinen experimentiert werden.
Die Klonierung ist eine molekularbiologische Methode, die dazu verwendet wird, bestimmte DNA-Abschnitte aus einem Organismus zu isolieren und in ein fremdes DNA-Molekül zu integrieren, um sie dann mithilfe von Wirtszellen zu vervielfältigen. Die einzubringende DNA wird dabei als Insert bezeichnet und das Plasmid als Vektor. Vektoren dienen als Transportmittel zur Übertragung der DNA-Sequenz (Insert) in eine Empfänger- bzw. Wirtszelle. Um das Insert in den Vektor einzubringen, kann die PCR-Methode (eine Polymerase-Ketten-Reaktion) angewendet werden. Restriktionsenzyme schneiden den Vektor und bilden so eine „Lücke“, in welche das Insert eingebracht wird.
Das Enzym Ligase verknüpft den Vektor und das Insert, sodass wieder ein geschlossener Vektor vorliegt. Der so entstandene rekombinierte Vektor kann dann weiterverarbeitet werden. Gemeinsam mit Anja habe ich eine Klonierung eines Plasmides durchgeführt. Dabei verwendeten wir zwei verschiedene Vektoren (pACEBac-1-His-tev, pACEBac-1-Sumo-tev) und ein Insert (Caspase 4). Nachdem wir unsere Ansätze vorbereitet hatten, konnte die DNA mittels der PCR vervielfältigt werden. Anschließend durfte ich durch die Agarose-Gelelektrophorese überprüfen, ob die PCR erfolgreich war. Durch den Anschluss einer Stromquelle wandern die eingefüllten Proben über das Gel und es erfolgt eine Auftrennung entsprechend der Größe. Durch die Verwendung eines Markers lassen sich die entstandenen Banden ablesen und können mit der Literatur auf die richtige Größe kontrolliert werden.
Unsere PCR war erfolgreich (1300 Basenpaare) und wir konnten mit den Produkten weiterarbeiten. Dafür haben wir diese aus dem Gel ausgeschnitten und aufgereinigt. Daraufhin fand der Restriktionsverdau und die Ligation statt.
Als nächsten Schritt führten wir eine „Heat-Shock-Transformation“ durch, bei welcher es unser Ziel war, dass die verwendeten kompetenten E.coli-Bakterien den rekombinierten Vektor aufnahmen. Dazu erhitzten wir beide Ansätze und kühlten diese anschließend auf Eis. Die durch Zentrifugieren gewonnenen Bakterienpellets wurden resuspendiert und auf Agar-Platten ausgestrichen, welche über Nacht bei 37 Grad inkubierten.
Auf den Agar-Platten befand sich auch ein Antibiotikum. Dieses dient der Selektion bzw. um zu erkennen, welche Bakterien den Vektor aufgenommen haben. Denn dieser enthält ein Gen, welches den Bakterien eine Antibiotikaresistenz verleiht. Es überlebten nur die Bakterien, die den Vektor aufgenommen hatten. Am nächsten Tag haben wir die Mini-Prep vorbereitet. Dazu pickten wir die Bakterienkolonien mit einer Pipette und gaben diese in ein Medium, welches die Bakterien mit Nährstoffen versorgte. Insgesamt hatten wir acht Proben, jeweils vier pro Vektor. Mithilfe des NanoDrop maßen wir die Konzentration der DNA, welche zwischen 38-154 µg/ml lag. Die Proben wurden über Nacht auf dem Schüttler inkubiert.
Die inkubierten Proben konnten dann erneut zentrifugiert werden und die dadurch gewonnenen Pellets wurden wieder resuspendiert und aufgereinigt. Das Endprodukt haben wir erneut auf ein Agarose-Gel aufgetragen. Dabei mussten wir feststellen, dass von unseren acht Proben nur drei mit der erforderlichen Bandengröße übereinstimmten. Die drei „positiven“ Proben haben wir dann zum Sequenzieren weggeschickt. Bei der Sequenzierung wird die Reihenfolge der Basen innerhalb der DNA bestimmt. Mittels der Sequenzierung wird überprüft, ob die Klonierung erfolgreich war und die Vektoren das Insert vollständig aufgenommen haben. Die drei eingeschickten Proben enthielten jedoch nicht die gewünschte Basenreihenfolge. Daher entschied sich Anja dafür, die gesamte Klonierung in der zweiten Woche noch einmal durchzuführen.
Das zweite große Projekt, an dem ich mitgearbeitet habe, war die Proteinexpression in E.coli-Bakterien und anschließende Proteinreinigung eines kleinen Antikörpers, eines sogenannten Nanobodys. Betreut wurde ich dabei von Martin, welcher als Doktorand im Institut arbeitet.
Die Expression dient der Gewinnung des modifizierten Proteins. Dafür wird ein Expressionssystem benötigt. Als solches wird jedes biologische System definiert, welches in der Lage ist, gezielt und kontrolliert eine Proteinbiosynthese zu betreiben. Dabei werden Proteine nach Vorlage der DNA hergestellt bzw. exprimiert. Alle lebenden Zellen stellen Expressionssysteme dar. Um möglichst viel Protein zu gewinnen, wird eine gezielte Überexpression angestrebt. Diese wird durch die Zugabe eines Induktors induziert.
Die Überexpression von E.coli-Bakterien wird durch ein Lac-Operon in Verbindung mit IPTG (Isopropyl-β-D-thiogalactopyranosid) erzielt. Die dadurch gewonnene Menge an modifiziertem Protein kann verwendet werden, um in weiteren Experimenten dessen Funktion oder Struktur zu untersuchen.
Für unsere Expression verwendeten wir E.coli-Bakterien als Startkultur, welche sich über Nacht in einem Medium samt passendem Antibiotikum bei 30°C im Inkubator vermehrten Zuvor hatten wir in regelmäßigen Abständen die Zellkonzentration mit dem NanoDrop gemessen und bei der Konzentration von 0,85 OD, welche nach ca. zwei Stunden erreicht war, den Induktor hinzugegeben.
Insgesamt stellen wir vier Ansätze her, je zweimal Nanobody X und Nanobody 7. Am nächsten Tag konnten wir die Bakterien ernten. Dazu wurden die Bakterienkulturen zentrifugiert und anschließend mit einem Spatel in einen Falcon gefüllt. Die entstandenen Pellets haben wir dann gewogen. Vom Nanobody X hatten wir insgesamt ca. 80g und vom Nanobody 7 ca. 35g gewonnen. Als die Expression vollständig durchlaufen war, musste das exprimierte Protein noch aufgereinigt werden.
Proteinaufreinigung kennzeichnet den Vorgang, bei welchem aus einem komplexen biologischen Gemisch (Zellkulturen) oder einer Lösung, ein oder mehrere Proteine herausgefiltert werden. Die Aufreinigung eines Proteins kann auf verschiedene Weisen erfolgen, z.B. durch die sogenannte Affinitätschromatographie.
Bei dieser handelt es sich um einchromatographisches Trennverfahren. Dazu werden ein geeigneter Ligand (Bindungspartner) und ein Analyt (Protein) benötigt. So ist es möglich, das gewünschte Protein zu isolieren. Die Affinitätschromatographie ist eine der leistungsfähigsten Trennmethoden. Die Trennung erfolgt meist durch Säulen. Das Protein besitzt eine Affinität gegenüber der Säule (stationäre Phase) und bindet an diese. Die restlichen Bestandteile der Lösung, wie Zellreste etc., werden einfach durchgespült. Der Durchfluss wird anschließend entsorgt. Erst wenn es zur Elution kommt, wird das Protein wieder von der Säule gelöst. Dies gewährleistet eine effektive Reinigung.
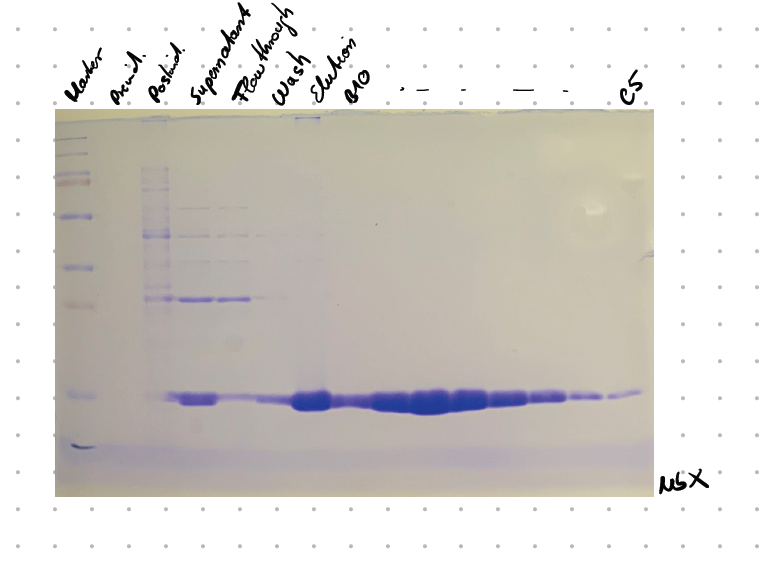
Für die Aufreinigung der Nanobodys haben wir Nickelbeats als Säulen verwendet. Zuerst mussten wir die festen Pellets mit TES Buffer resuspendieren und über Nacht in einem Kälteraum lagern. Am nächsten Morgen filterten wir die Proben und ließen sie anschließend über die Säulen laufen. Die Säulen wurden vorher mit dem Wash Buffer gewaschen und der Durchfluss entsorgt.
Nachdem das gesamte Protein auf die Säule aufgetragen war und der Durchfluss ebenfalls entsorgt war, konnten wir mit dem Elution Buffer das Protein wieder von den Nickelbeats lösen und in Falcons sammeln.
Die so entstandenen Proteinlösungen verwendeten wir dann für eine Gelfiltration. Bei dieser Variante der Chromatographie werden die Moleküle nach ihrer Größe und Form aufgetrennt.
Die Grundlage einer solchen Gelfiltration ist eine mit einer speziellen Gelmatrix gefüllte Säule. Die Proteinlösung wird auf die Säule gegeben. Aufgrund der Beschaffenheit der Säule wandern große Moleküle schneller über diese, da sie schlechter in die Poren der Gelmatrix eindringen können als kleinere Moleküle, welche langsamer durch die Matrix gelangen. Je kleiner ein Molekül also ist, desto länger dauert es, bis es die Säule durchlaufen hat. Die durchgelaufene Proteinlösung wird in Fraktionen eingeteilt und gesammelt. Der Computer detektiert diese und wertet sie aus. Der Nanobody 7 wurde in den Fraktionen B12-C7 und der Nanobody X in B10-C5 detektiert.
Den Inhalt der Fraktionen haben wir dann in Eppiröhrchen gesammelt und auf ein selbst gegossenes SDS-Gel aufgetragen, um zu überprüfen, ob das Protein die richtige Bandengröße hatte. Die SDS-PAGE ist eine Variante der Polyacrylamid-Gelelektrophorese, bei welcher es sich um eine analytische Methode der Biochemie zur Trennung von Stoffgemischen nach der Molekülmasse in einem elektrischen Feld handelt. Mit dieser Methode lassen sich Proteine mit Molekülmassen zwischen 5 und 250 kDa auftrennen. Die Wanderungsgeschwindigkeit im Gel ist von der Molekülgröße abhängig. Je kleiner ein Protein ist, desto schneller wandert es im Gel. Das SDS-Gel besteht aus einem Sammel- und einem Trenngel. Im Sammelgel werden die Proteine aus der Probenlösung konzentriert, bevor sie im Trenngel separiert werden. Durch das Aufkonzentrieren wird die anschließende Bandenbildung gefördert. Die Blaufärbung entsteht durch die Verwendung des Farbstoffs Coomassie. Die beiden Nanobodys hatten eine Größe von 15 kDa. Anschließend haben wir die Proteinproben mithilfe eines Spezialbehälters (Amicon) aufkonzentriert und dann in flüssigem Stickstoff eingefroren. Neben der Proteinherstellung und Aufreinigung hatte ich außerdem die Möglichkeit bei einem biochemischen und biophysikalischen Experiment mitzuarbeiten. Dabei durfte ich verschiedene technische Geräte kennenlernen, die Proteinbindungen messen.
Gemeinsam mit Michael konnte ich mit dem MST (Microscale Thermophoresis) arbeiten. Dabei wird die gerichtete Bewegung von Molekülen entlang eines Temperaturgradienten gemessen. Dieser Vorgang wird als Thermophorese bezeichnet.
Die Thermophorese ist von vielen Faktoren abhängig, die sich bei Bindung eines Liganden verändern. Für die Messung wird das zu untersuchende Protein fluoreszenzmarkiert. Die Thermophorese wird nach lokaler Erhitzung durch einen Infrarotlaser optisch erfasst. Die Veränderungen werden in Form eines Diagramms wiedergegeben. Anhand dieser lassen sich die Bindungsparameter quantitativ bestimmen.
Zusammengefasst lässt sich sagen, dass ich ein sehr spannendes Praktikum absolvieren konnte, bei welchem ich Einblicke in die molekularbiologischen, biochemischen und biophysikalischen Arbeiten des Instituts bekommen konnte.
Ich würde jedem, der sich für die Bereiche Biochemie und Forschung interessiert, dieses Praktikum unbedingt empfehlen. Es ist eine einzigartige Möglichkeit, um einen realistischen Eindruck von der Laborarbeit zu gewinnen.
Außerdem möchte ich mich bei dem gesamten Team des Instituts für Strukturbiologie bedanken, die mich jederzeit sehr gut betreut und unterstützt haben. Ein ganz besonderer Dank gilt auch den Auricher Wissenschaftstagen, die das Praktikum und die damit einhergegangenen Erfahrungen erst ermöglicht haben.