Von Amir Charafeddine und Aliena Petretto
06. bis 18. Oktober 2024


Im Rahmen der Auricher Wissenschaftstage hatten wir – Amir Charafeddine und Aliena Petretto – die besondere Möglichkeit, vom 6. bis 18. Oktober ein zweiwöchiges Praktikum am Institute of Structural Biology in Bonn zu absolvieren.
Das Institute of Structural Biology bietet Forschungsmöglichkeiten im Bereich der Strukturbiologie mit Fokus auf der biochemischen Analyse zellulärer Regulationsprozesse. Hierbei wirdeine Vielzahl von Techniken aus der Molekularbiologie und Biochemie verwendet, um Proteine, Protein-Wechselwirkungen sowie die Wechselwirkung mit Lipiden, Modellmembranen, Nukleinsäuren und kleinen molekularen Liganden zu bestimmen.
Während unseres Aufenthalts am Forschungsinstitut wurden wir von den Mitgliedern der Arbeitsgruppe unter der Leitung von Prof. Dr. Matthias Geyer betreut und durch die zwei Wochen unseres Praktikums begleitet.
Unser erster Tag am Institute of Structural Biology startete morgens um neun mit einem „Labmeeting“. Hierbei handelte es sich um ein Onlinemeeting, bei dem wichtige organisatorische Fragen und Ankündigungen für die kommende Woche besprochen wurden. Bei der Gelegenheit wurden wir als Praktikanten der Auricher Wissenschaftstage dem Team vorgestellt.
Nach dem Meeting erhielten wir eine Führung durch das Institut, um die Räumlichkeiten kennenzulernen und uns mit ihnen vertraut zu machen.
Im Anschluss daran wurde uns dann unsere Aufgabe für die nächsten Tage mitgeteilt, ein Laborpraktikum, welches für drei Tage ausgelegt ist. Christoph Winterberg erklärte uns dafür die Grundlagen, um die auf uns zu dem Zeitpunkt hochkomplex wirkenden Prozesse zu verstehen.
Das Ziel dieses Laborpraktikums war es, die Expression und Reinigung des Proteins TEV-Protease in Escherichia coli (E. coli) zu untersuchen. Die TEV-Protease ist eine Cysteinprotease, die allgemein als 27 kDa große katalytische Domäne des Nuclear Inclusion a (NIa) Proteins bekannt ist, welches vom Tabakmosaikvirus (Tobacco Etch Virus, TEV) kodiert wird. Durch das Praktikum sollten wir Einblicke in die biochemischen und molekularbiologischen Techniken erhalten, die zur Proteinexpression und -reinigung verwendet werden. Die folgenden Schritte beschreiben das Vorgehen bei der Expression der TEV-Protease in E. coli, der Isolierung des Zielproteins aus den E. coli-Zellen und der anschließenden Reinigung mittels Affinitäts- und Größenausschlusschromatographie. Schließlich wurde die Aktivität der TEV-Protease unter verschiedenen Bedingungen (Temperatur und Konzentration) getestet.
Der erste Schritt, die Proteinexpression aus der Plasmid-DNA, wurde bereits vor unserer Ankunft erledigt. Wir haben jedoch dieselben Zellkultivierungs- und Expressionsvorgänge unter der Aufsicht von Maria Zyulina später selbst durchgeführt. Um einen stringenten und kohärenten Ablauf zu gewährleisten, steht dieser Vorgang nun trotzdem am Anfang.
Als Vorbereitung wurde die Expression der TEV-Protease in E. coli-Zellen vorbereitet. Die Expression eines Proteins in E. coli ermöglicht die effiziente Produktion großer Mengen des gewünschten Proteins. Dieser Prozess beginnt mit der Vorbereitung einer 1-Liter-Kultur im LB-Medium (ein komplexes Nährmedium zur Kultivierung von Bakterien), das mit Ampicillin ergänzt wurde, um sicherzustellen, dass nur Bakterien wachsen, die das Plasmid mit dem TEV-Gen tragen.
Nach der Inokulation der Hauptkultur mit der Übernachtkultur wurde diese bei 37°C und unter ständigem Schütteln inkubiert, um das Bakterienwachstum zu fördern. Das Wachstum der Kultur wurde regelmäßig bei einer Wellenlänge von 600 nm (OD600) gemessen, um die Zellkonzentration zu überwachen. OD600 ist eine Abkürzung zur Angabe der optischen Dichte einer Probe, gemessen bei einer Wellenlänge von 600 nm in 1 cmLichtweg. Als die Kultur eine OD600 von 0,7 erreichte, wurde die Expression der TEV-Protease durch Zugabe von 0,5 Milimolar (mM) IPTG induziert. IPTG ist ein Molekül, das den Promotor des Plasmids aktiviert und somit die Produktion der TEV-Protease startet.
Nach der Induktion wurde die Kultur für weitere drei Stunden bei 22°C inkubiert, um eine optimale Expression des Proteins zu gewährleisten, ohne dass die Zellen überbelastet werden. Die reduzierte Temperatur verringert die Bildung von Einschlusskörpern und fördert die Produktion von löslichem Protein. Anschließend wurde die Kultur zentrifugiert, um die Zellen vom Medium zu trennen. Das resultierende Zellpellet wurde gewogen und bei -80°C eingefroren, um die Zellen für die nachfolgende Reinigung zu konservieren.
Parallel zur Expression wurden Pufferlösungen für die Reinigung der TEV-Protease vorbereitet. Diese Puffer sind essenziell, um das Protein zu stabilisieren und bei der Reinigung spezifisch zu isolieren.


Als nächster Schritt im Rahmen des Laborpraktikums wurde die Zelllyse und Reinigung der TEV-Protease durchgeführt. Dieser Prozess ist entscheidend, um das Zielprotein von anderen zellulären Bestandteilen zu trennen und eine reine Proteinzubereitung zu erhalten.
Die Zellen wurden mit einem Sonikator aufgebrochen, um das Protein aus den Zellen zu extrahieren. Der Sonikator verwendet hochfrequente Schallwellen, um die Zellmembranen zu durchbrechen, wodurch die intrazellulären Komponenten freigesetzt werden. Das Zellpellet wurde im 5-fachem Volumen seines Gewichts in Bindepuffer resuspendiert und zweimal sonifiziert (40 % Ampl., 5 s Puls, 10 s Pause). Die Zelltrümmer und unlöslichen Bestandteile wurden durch Zentrifugation entfernt. Der resultierende Überstand, der das gelöste Protein enthielt, wurde für die Affinitätschromatographie verwendet.
Die Reinigung der TEV-Protease erfolgte dann mittels Affinitätschromatographie. Dabei wurde das Protein durch seinen His-Tag an eine Ni²⁺-beladene Agarose-Säule gebunden. Der His-Tag besteht aus sechs Histidinresten, die stark an das Nickel im Säulenmaterial binden, was die spezifische Isolation des Zielproteins ermöglicht. Der Überstand wurde mit den vorbereiteten Beads, winzige kugelförmige Partikel, gemischt und inkubiert, damit das Protein an die Beads binden konnte. Anschließend wurde die proteinhaltige Bead-Suspension in eine Säule überführt und mehrfach gewaschen, um nicht gebundene Proteine zu entfernen.

Die abschließende Gelfiltration diente dazu, das Protein weiter zu reinigen und unerwünschte kleine Moleküle wie Imidazol zu entfernen, die während der Affinitätschromatographie verwendet wurden. Die gelierte Proteinlösung wurde auf einer Superdex 75 10/300 GL-Säule aufgetragen und die Absorption bei 280 Nanometer (nm) überwacht. Die gelösten Fraktionen, die das Protein enthielten, wurden kombiniert und ihre Konzentration mittels NanoDrop bei 280 nm bestimmt.Das NanoDrop bietet eine Messung der Absorption von etwa 200 nm bis 350 nm, was dem relevanten Bereich zum Bestimmen der Konzentration und Reinheit von RNA/DNA entspricht.
Schließlich wurde das Protein auf eine Konzentration von 3 mg/ml eingestellt, in flüssigem Stickstoff schockgefroren und bei -80°C gelagert.
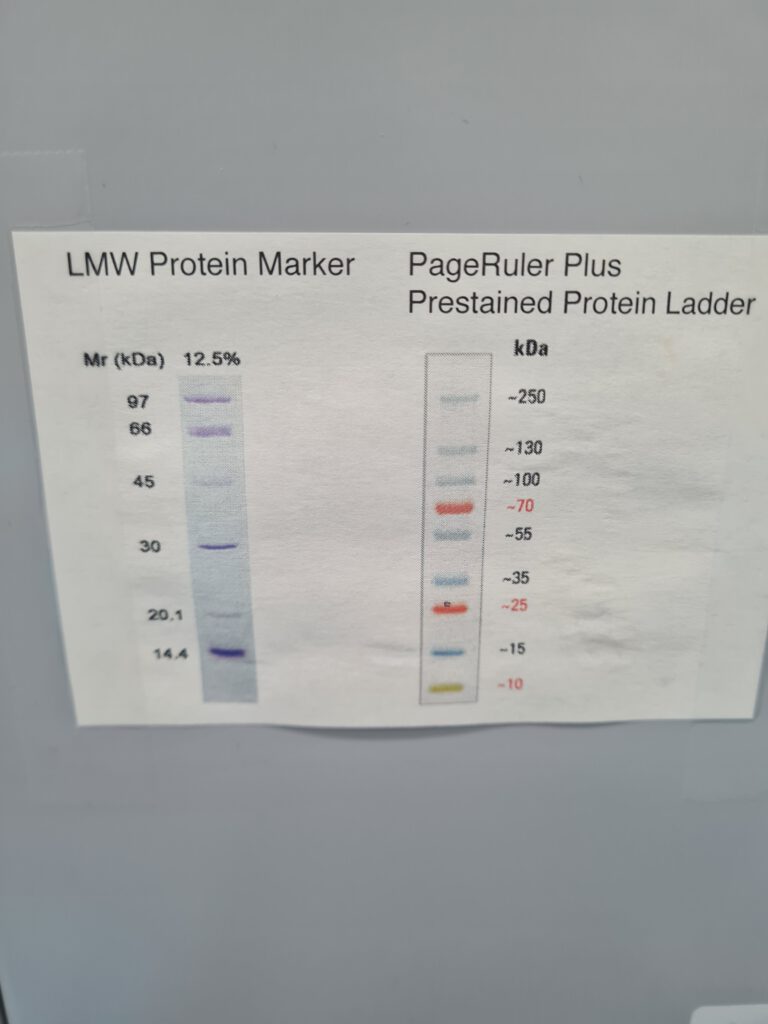
In den letzten Schritten des Laborpraktikums wurden die gereinigten Proteinproben mittels SDS-PAGE analysiert, um die Reinheit und Größe der Proteine zu überprüfen. Die SDS-PAGE ist eine Methode, bei der Proteine nach ihrer Größe getrennt werden, indem sie in einem polyakrylamidgefüllten Gel in Anwesenheit von SDS wandern. Dieser Schritt ist wichtig, um die unterschiedlichen Reinigungsschritte der TEV-Protease visuell zu bestätigen. Das Gel wurde bei 175 V für 50 Minuten laufen gelassen. Danach wurden die Proben mit einem Coomassie-Brilliant-Blue-Färbebad gefärbt und mit destilliertem Wasser entfärbt, um die Proteinbanden sichtbar zu machen.
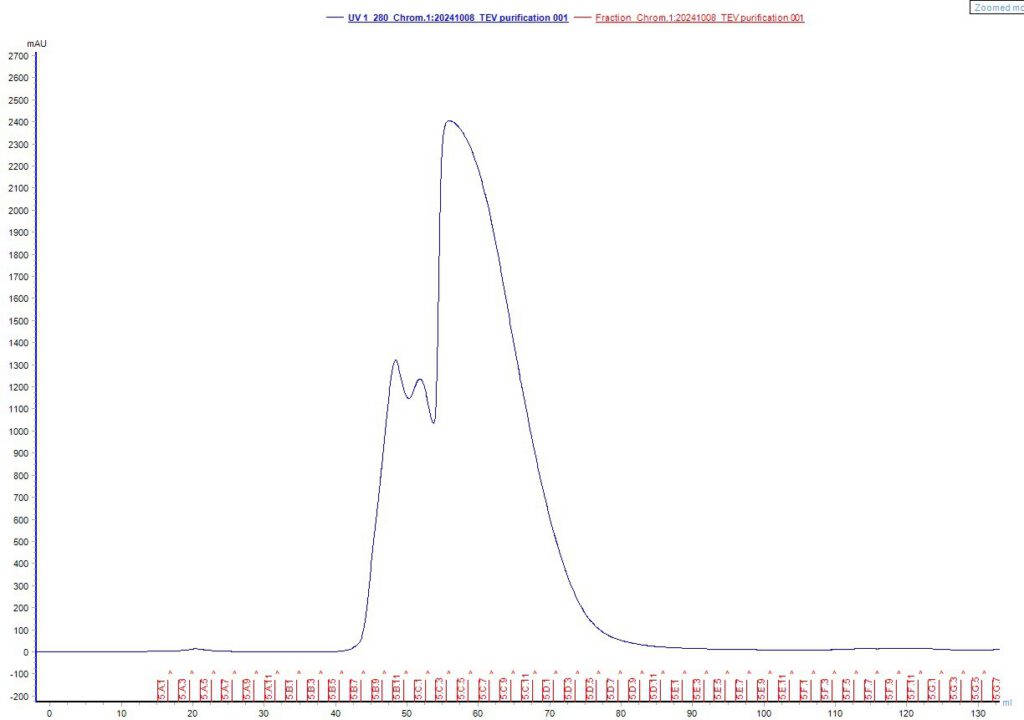

Parallel dazu führten wir einen activity assay durch, um die Funktionalität der gereinigten TEV-Protease zu testen, in diesem Fall wie gut die TEV-Protease das Substratprotein an der Erkennungsstelle spaltet. TEV-Protease erkennt ein spezifisches lineares Epitop und schneidet zwischen den Aminosäuren Q (Glutamin) und G (Glycin) oder Q und S(Serin). Der Assay nutzte ein Substratprotein mit dieser Erkennungssequenz. Während des Assays wurden verschiedene Parameter wie Zeit, Temperatur und Enzym-Substrat-Verhältnisvariiert, um optimale Bedingungen für die Proteaseaktivität zu ermitteln.
Die Ergebnisse der SDS-PAGE und des activity assay zeigten, dass die TEV-Protease erfolgreich exprimiert und gereinigt wurde und eine hohe Aktivität aufwies. Die Reinheit der Proteine und die Effizienz der Proteolyse wurden durch diese Experimente bestätigt.Diese Daten sind entscheidend, um sicherzustellen, dass die Protease in nachfolgenden Anwendungen zuverlässig funktioniert, beispielsweise bei der Entfernung von Fusions-Tags aus rekombinanten Proteinen.
Während unseres Praktikums hatten wir neben der Arbeit im Labor außerdem die Möglichkeit, zwei spannendeVorträge zu besuchen. Sophie Binder sowie Prof. Malcom White aus Schottland referierten unter anderem über neue Ergebnisse im Bereich desCRISPR/Cas-Systems. CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) bezeichnet spezifische DNA-Sequenzen, die im Erbgut vieler Bakterien und Archaeen vorkommen. Diese Abschnitte bestehen aus sich wiederholenden Sequenzen (Repeats) und spielen eine zentrale Rolle im Abwehrmechanismus dieser Mikroorganismen. Gemeinsam mit den sogenannten Cas-Proteinen (CRISPR-associated Proteins) bildet das CRISPR/Cas-System eine Art adaptives Immunsystem, welches die Zellen vor fremdem genetischem Material, wie es von Viren oder Plasmiden stammt, schützt. Durch diesen Mechanismus können Prokaryoten fremde DNA erkennen, zerschneiden und so eine erneute Infektion verhindern. Das System speichert zudem Bruchstücke des fremden Erbguts, um zukünftige Angriffe schneller und gezielter abwehren zu können. Die molekularbiologische Forschung hat das CRISPR/Cas-System als Grundlage für eine präzise und vielseitige Methode zur Genomeditierung nutzbar gemacht. Mit dieser Technik lassen sich gezielt Veränderungen in der DNA von Organismen vornehmen, was in der Biotechnologie, Medizin und Landwirtschaft neue Möglichkeiten eröffnet.
Des Weiteren durften wir unter anderem Dominic Ferber bei der Arbeit mit verschiedenenMessgeräten begleiten. Hierbei erklärte er uns die Funktionsweise der verschiedenen Messmethoden, welche in der Strukturbiologie angewandt werden. Eine dieser Methoden ist die sogenannte Oberflächenplasmonenresonanz, auf Englisch „Surface Plasmon Resonance“ (kurz SPR). Hierbei handelt es sich um eine optische Methode, die zur Messung molekularer Interaktionen in Echtzeit eingesetzt wird. SPR tritt auf, wenn linear polarisiertes Licht unter den Bedingungen der totalen internen Reflexion auf einen Metallfilm trifft.
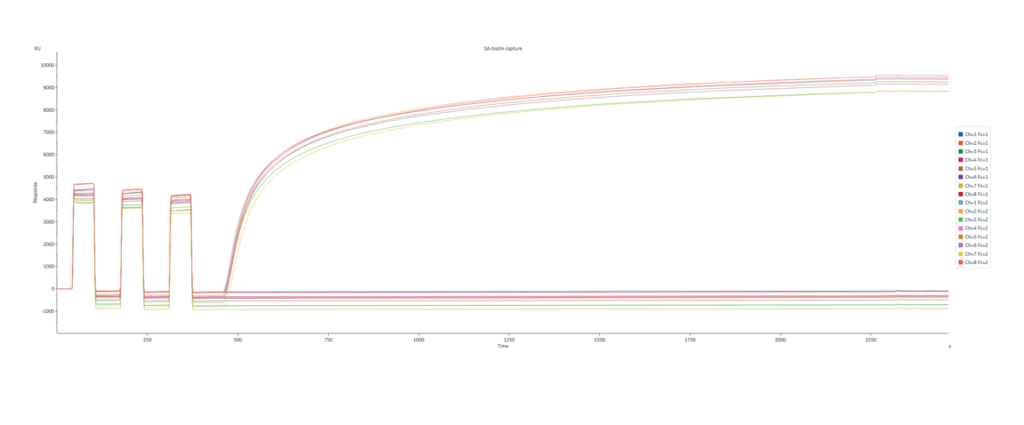
Das SPR-Signal hängt direkt vom Brechungsindex des Mediums auf dem Sensorchip ab. Die Bindung von Biomolekülen bewirkt Veränderungen im Brechungsindex auf der Sensoroberfläche.Während eines SPR-Experiments wird ein Molekül (der Ligand) auf einem Sensorchip immobilisiert und die Bindung eines zweiten Moleküls (der Analyte) wird unter kontinuierlichem Fluss einer Pufferlösung gemessen. Die Reaktion wird in Resonanzeinheiten (RU) ausgedrückt, die proportional zur Masse auf der Sensoroberfläche sind. Für eine spezifische Interaktion ist die gemessene Reaktion proportional zur Anzahl der Moleküle, die an die Oberfläche gebunden sind. Die Ergebnisse werden in Echtzeit erfasst und als Sensogramm dargestellt.
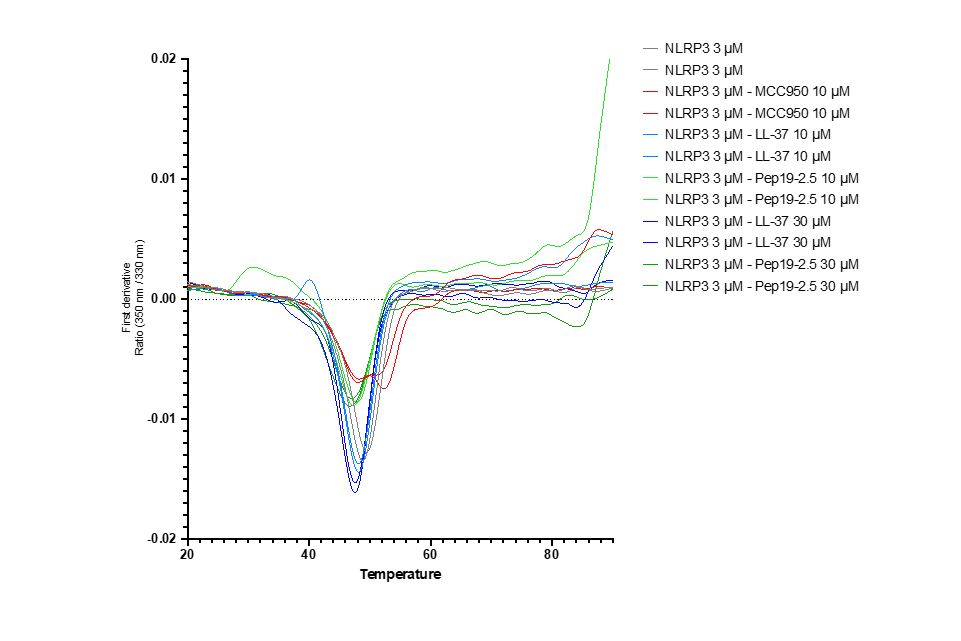
Die zweite Methode, die nanoDSF(differential scanning fluorimetry) ist eine biophysikalische Methode zur Bewertung der konformationellen Stabilität von Proteinen, alsowie widerstandsfähig das Protein gegenüber Veränderungen seiner dreidimensionalen Struktur (Konformation)ist. Sie untersucht, wie wahrscheinlich es ist, dass ein Protein unter stressvollen Bedingungen gefaltet und funktional bleibt. Dies geschieht durch die Überwachung der intrinsischen Fluoreszenz eines Proteins, während es thermischem oder chemischem Stress ausgesetzt wird. Falls das Protein unter diesem Stress entfaltet wird, ändern sich seine fluoreszierenden Eigenschaften. Der Punkt, an dem die Entfaltung eintritt (die sogenannte „Schmelztemperatur“ oder Tm), gibt Aufschluss über die allgemeine Stabilität der Proteinstruktur.
Die nanoDSF ist äußerst empfindlich und kann selbst kleinste Veränderungen in der Fluoreszenz eines Proteins erkennen, wodurch ein detailliertes Bild seiner konformationellen Stabilität entsteht. Sie ermöglicht die Messung sowohl der thermischen Stabilität (Verhalten des Proteins bei steigenden Temperaturen) als auch der chemischen Stabilität (Verhalten in Gegenwart von denaturierenden Chemikalien). Die Methode liefert umfangreiche Daten, darunter die Temperatur, bei der die Hälfte des Proteins entfaltet ist, die Temperatur, bei der die Entfaltung beginnt, die Steigung der Entfaltungskurve sowie die Aktivierungsenergie der Entfaltung.
Diese Parameter sind entscheidend für das Verständnis der thermodynamischen Eigenschaften eines Proteins und dienen als Schlüsselfaktororen für seine Funktion und Anwendung.
Darüber hinaus kann die nanoDSF auch geringste Änderungen in der konformationellen Stabilität eines Proteins erfassen, die durch die Bindung eines Liganden verursacht werden. Dies ermöglicht ein effizientes Hochdurchsatz-Screening von Ligandenbindungen, beispielsweise im Rahmen eines Thermal Shift Assay (TSA).
Während der letzten Tage unseres Praktikums durften wir Dr. Nicola Willemsen und Hannes Buthmann bei der Arbeit mit Insektenzellen und menschlichen Zellen über die Schulter schauen.

Nachdem die zwei Wochen am Institute of Structural Biology wie im Flug vergangen sind, hieß es dann Abschied nehmen, um am Freitagnachmittag mit dem Zug zurück nach Aurich zu fahren.
Zum Abschluss möchten wir den Auricher Wissenschaftstage unseren herzlichen Dank aussprechen, die es uns ermöglicht haben, das Praktikum am Institute of Structural Biology in Bonn zu absolvieren.In den zwei Wochen, die wir dort verbringen durften, haben wir viel über den Bereich der Strukturbiologie gelernt und hatten, die Möglichkeit neueErfahrungen – vor allem im fachpraktischen Bereich- zu sammeln. Außerdem hatten wir das Glück, viele nette Leute kennenzulernen, die uns jederzeit bei unserer Arbeit unterstützt habenund sich die Zeit dafür nahmen, uns unsere Fragen zu beantworten und uns mit Erzählungen aus eigener Erfahrung viele wertvolle Tipps für unser kommendes Studium und unseren Werdegang gegeben haben.
An dieser Stelle möchten wir uns ganz herzlich beim Team des Institute of Structural Biology für die tolle Zeit bedanken, insbesondere bei Prof. Dr. Matthias Geyer, der das Praktikum in Zusammenarbeit mit den Auricher Wissenschaftstagen erst möglich gemacht hat und bei Christoph Winterberg, Dr. Nicola Willemsen, Dominic Ferber, Hannes Buthmann und Maria Zyulina für die Betreuung während unserer Zeit dort.










